陈柱成实验室
Chen Zhucheng Laboratory
Ye Y#, Wu H#, Chen K, Clapier CR, Verma N, Zhang W, Deng H, Cairns BR*, Gao N*, Chen Z*. Structure of the RSC complex bound to the nucleosome. Science, 366(6467):838-843, 2019.
https://www.science.org/doi/epdf/10.1126/science.aay0033
新闻推送
https://www.life.tsinghua.edu.cn/info/1131/2181.htm
2019年11月1日,清华大学生命学院陈柱成、北京大学生命学院高宁与美国犹他大学Bradley Cairns课题组在《科学》(Science)杂志上在线发表题为 《核小体结合状态的染色质重塑复合物RSC的结构》(Structure of the RSC complex bound to the nucleosome)的研究论文。该工作解析了RSC-核小体复合物的高分辨冷冻电镜结构(整体分辨率7.1埃,核心区域3.0埃),揭示了RSC复合物对染色质的重塑机理。
染色质是遗传信息的物质载体。染色质的形成可以有效压缩DNA以适应细胞空间的需要,但同时抑制了基因的表达。核小体是组成各级染色质结构的基本单位。染色质重塑复合物(Chromatin remodeler)改变核小体位置和组成,对染色质结构进行重塑,在基因表达、DNA复制与修复等过程中发挥着重要作用。RSC是酵母细胞的关键染色质重塑复合物,调控酵母大部分基因的表达。RSC复合物包含15个亚基,其分子量在1MDa以上。尽管被广泛研究,RSC如何组装,如何滑动核小体,打开基因启动子的机理仍有赖于进一步阐释。
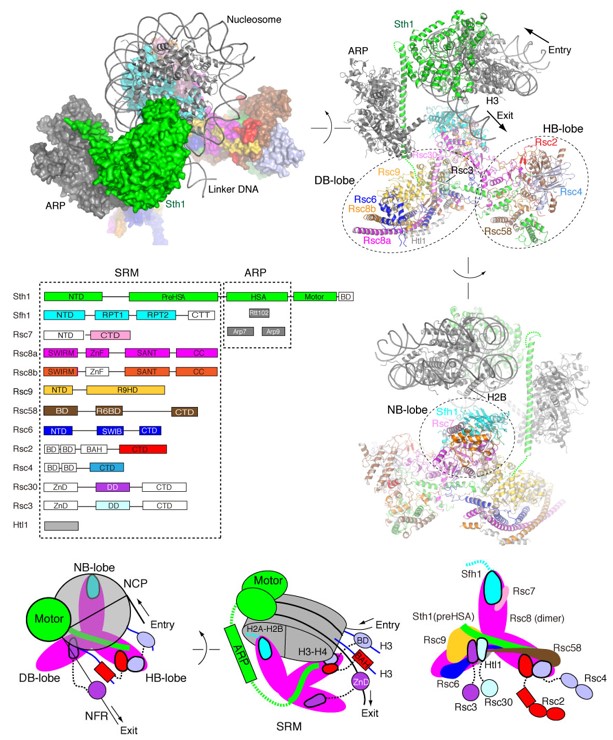
图1 RSC-核小体复合物的整体结构以及核小体识别模型
陈柱成实验室自建立起,对染色质重塑过程进行了系列深耕。曾经报道染色质重塑蛋白Snf2和ISWI在不同状态下的结构 (Yan, Nature 2016; Liu, Nature 2017; Li, Nature 2019)。在这些研究基础上,陈柱成与高宁、Bradley Cairns实验室合作,解析了RSC-核小体复合物的高分辨结构,并对关键亚基进行了功能研究。该结构显示,RSC分为三个功能模块(图1),即马达模块(Motor)、ARP模块与底物招募模块(SRM),这三个模块由核心亚基Sth1串联组成。与前期普遍认为的“空腔-嵌入”模型不同,该工作发现RSC主要通过马达与核小体相互作用,这支持了统一的ATP依赖的染色质重塑模型。ARP模块主要起到支架和调控马达的作用。SRM包含三个底物招募叶片:DNA结合叶片(DB-lobe)、组蛋白尾肽结合叶片(HB-lobe)与核小体结合叶片(NB-lobe)。这些叶片在空间上被安置在它们对应的底物附近,使它们能够协同作用,结合核小体。这个工作阐明了RSC识别启动子的染色质特征,定向移动核小体,打开启动子的机理。RSC的多个亚基在生物进化过程中高度保守,RSC结构的解析为理解人源复合物(PBAF)提供了模版。其中,在NB-lobe的Sfh1亚基被发现结合组蛋白H2A-H2B酸性区表面,促进了RSC在体内和体外的染色质重塑活性,这提示Sfh1的同源蛋白INI1/ BAF47/SMARCB1突变造成多种癌症和神经发育疾病的可能机理。

图2 团队略影
清华大学2015级博士生叶佑丕(陈柱成课题组)、2014级博士吴昊(高宁组)为本文共同第一作者,博士生陈康净(陈柱成课题组)、张文浩(邓海腾课题组)参与了重要工作(图2)。清华大学生命学院陈柱成教授、北京大学生命学院高宁教授以及美国犹他大学Bradley Cains教授为本文共同通讯作者。本课题由中国科技部,国家自然科学基金委提供经费支持,并得到清华-北大生命科学联合中心,北京市高精尖结构生物学创新中心的资助。
全文链接:https://science.sciencemag.org/content/early/2019/10/30/science.aay0033
相关文献:
https://www.nature.com/articles/nature20590
https://www.nature.com/articles/nature22036
https://www.nature.com/articles/s41586-019-1029-2

扫一扫关注微信